Démarrer avec R et découverte de RStudio
Introduction
Ce tutoriel est composé de 4 parties :
- Partie 1 : présentation de R et de RStudio, gestion du répertoire de travail, des librairies, …
- Partie 2 : présentation des principaux objets en R, les premiers graphiques, la programmation, l’importation/exportation, …
- Partie 3 : présentation d’outils pour la manipulation des lois usuelles, de statistiques descriptives, …
- Partie 4 : présentation de tidyverse (en particulier de dplyr, stringr, ggplot2) et les essentiels de la rédaction de documents avec Rmarkdown.
La rédaction de ces tutoriels s’appuie principalement sur :
- des supports développés avec/par des collègues de l’INSA Toulouse et de l’IMT (qu’ils en soient ici remerciés)
- les livres suivants :
- R for data science: import, tidy, transform, visualize, and model data de Wickham, H. and Grolemund, G. (2016)
- Statistiques avec R de Cornillon et al. (2010)
- R markdown: The definitive guide de Xie et al. (2018)
- ggplot2: elegant graphics for data analysis de Wickham (2016)
- de plusieurs sites web qui seront mentionnés au fur et à mesure
A propos de R 
Le logiciel R est développé depuis les années 90 par un groupe de volontaires de différents pays et par une large communauté d’utilisateurs. C’est un logiciel libre, publié sous licence GNU GPL et multiplateforme (fonctionne sur systèmes Linux, Mac OS X ou Windows). Il est facile à installer à partir de la page du CRAN (ou d’un site miroir); le CRAN contient toutes les ressources nécessaires à l’utilisateur de R, débutant ou expérimenté : fichiers d’installation, mises à jour, librairies, FAQ, newsletter, documentation… R fait partie des logiciels les plus utilisés de la communauté statistique académique et aussi de plus en plus dans les services R&D des entreprises en concurrence avec les logiciels commerciaux.
Dans sa structure, R est un langage de programmation d’une syntaxe voisine à celle du langage C et capable de manipuler des objets complexes sous forme de matrice, scalaire, vecteur, liste, facteur et aussi data frame. Il dispose également d’une très riche librairie de quasiment toutes les procédures et méthodes statistiques de la littérature. Plus précisément, toutes les recherches récentes sont d’abord développées et diffusées à l’aide de ce logiciel par la communauté scientifique.
Pour suivre cette série de tutoriels, vous devez commencer par installer R (version > 4.0.1) sur votre ordinateur. Pour cela, rendez-vous sur l’une des pages suivantes et suivez les instructions :
A propos de RStudio 
RStudio n’est pas qu’une interface graphique pour R, il s’agit d’un environnement de développement intégré, proposant des outils et facilite l’écriture de scripts et l’utilisation de R. La version gratuite de RStudio étant déjà très riche en fonctionnalités, elle est suffisante pour les enseignements.
Description des fenêtres de RStudio
Une fois R et RStudio installés sur votre machine, vous pouvez
démarrer RStudio.
Au lancement de RStudio, l’écran est découpé en trois grandes
zones.
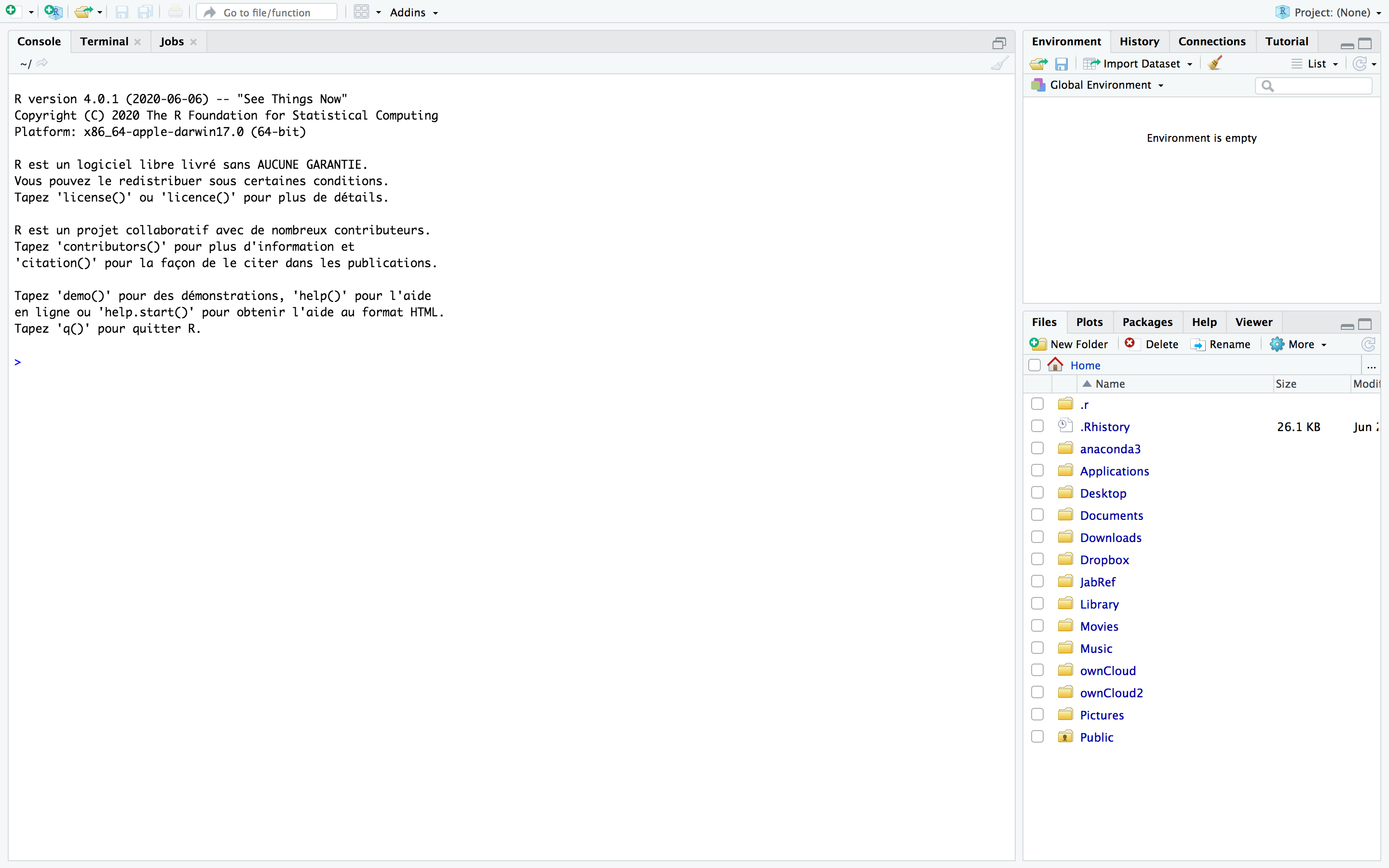
3 zones à l’ouverture de RStudio
La zone de gauche s’appelle la Console, on peut y
lire au lancement la version de R que RStudio a chargé. Après le texte
d’entrée, la ligne commence par > qui est l’invite de
commande. RStudio est prêt à recevoir votre première commande. On peut
se servir de R comme d’une calculatrice. Vous pouvez par exemple taper
> 2+3 et taper Entrée. Le résultat apparait
alors dans la console.
En haut à droite, on peut voir dans Environnement la liste des objets et fonctions créés au fur et à mesure du travail. On a également accès à l’historique des commandes dans History.
En bas à droite, on trouve plusieurs onglets :
- Files : permet de voir le contenu du répertoire de travail
- Plots : permet de visualiser les graphiques créés
- Packages : liste les packages installés pour les charger, mettre à jour, en installer d’autres (voir la section dédiée)
- Help : permet d’accéder à l’aide en ligne
Vous pouvez modifier la configuration des onglets de RStudio en
allant dans Preferences -> Pane Layout.
Types de documents avec RStudio
En allant dans File -> New File ou en cliquant sur la
flèche à côté de l’icone de New File (en haut à gauche),
RStudio propose de nombreux types de document. Vous pouvez noter que
l’on peut utiliser RStudio également pour faire du python, du C++, …
Nous allons ici ne parler que de quelques types de fichiers que l’on
utilisera par la suite. Lorsque l’on sélectionne un type de fichier, une
quatrième fenêtre en haut à gauche de l’interface de RStudio
s’ouvre.
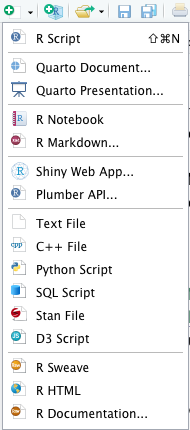
Les types de fichiers disponibles avec RStudio
Pour créer un script R, il suffit de sélectionner
R Script. Ce script peut être enregistré à tout moment dans
un fichier avec l’extension ” .R” (ex. monScript.R) dans le
répertoire courant en cliquant sur l’icône de disquette ou par
File->Save. Vous pouvez l’ouvrir à tout moment grâce au
menu File –> Open file..., à l’icône dossier ou en
double cliquant sur le fichier monScript.R. Il est également
possible d’executer un script directement depuis la console grâce à la
commande source("monScript.R").
Vous pouvez créer un document (rapport, slides, …) à l’aide de
RMarkdown en sélectionnant R markdown .... En particulier,
vous avez la possibilité d’éditer directement des documents au format
pdf ou html (en incluant les commandes R, les sorties et les graphes).
Le nouveau document créé sera sauvegardé avec l’extension ” .Rmd”. Lors
de la création, il faut préciser le type de document final souhaité
(document au format HTML ou PDF ou presentation, etc), ainsi que les
informations du document (titre et auteur). On reviendra plus
précisément sur les possibilités offertes par Rmarkdown et les commandes
principales pour débuter la rédaction d’un rapport dans le tutoriel 4 de
R avancé.
Environnement
Répertoire de travail
Pour pouvoir récupérer des données, maîtriser la sauvegarde des
scripts, des résultats, … il est important de connaitre le répertoire de
travail c’est-à-dire le répertoire sous lequel les divers résultats
seront sauvegardés par défaut. Ce dernier s’obtient à l’aide de la
commande getwd().
Pour changer de répertoire de travail, on peut utiliser la commande
setwd() dans la Console. A noter que R ne reconnaît que le
caractère “/” pour spécifier le chemin d’accès d’un répertoire (même
sous Windows). On peut aussi passer par
Session -> Set Working Directory -> Choose Directory.
L’aide de R
Il est normal de ne pas savoir (ou d’avoir oublié) quels sont les
arguments d’une fonction, ou comment ils se nomment. On peut à tout
moment faire appel à l’aide intégrée de R. On peut accéder à l’aide pour
une fonction (ex pour la fonction plot) en passant le nom de la
fonction dans help() (ex help(plot)) ou en
utilisant le raccourci ?plot. Ces deux commandes permettent
d’afficher une page de description (en anglais) de la fonction, ses
paramètres, son résultat, d’exemples, … Ces pages d’aide contiennent de
nombreuses informations mais elles ne sont pas toujours d’une lecture
aisée. Dans RStudio, les pages d’aide en ligne s’ouvriront par défaut
dans la zone en bas à droite, sous l’onglet Help. Un
clic sur l’icône en forme de maison vous affichera la page d’accueil de
l’aide. Vous pouvez aussi taper directement le nom de la fonction
recherchée dans la zone de recherche dans l’onglet
Help.
Les librairies pour R
R est un logiciel libre, il s’enrichit grâce au développement de packages par la communauté. La plupart des extensions sont développées et maintenues par la communauté des utilisateurs de R, et diffusées via un réseau de serveurs nommé CRAN (Comprehensive R Archive Network). On peut également trouver des packages R dédiés à l’étude des données génomiques à haut-débit sur Bioconductor, des packages déposés sur github, …
Pour installer une librairie disponible sur le CRAN, vous avez plusieurs manières de le faire :
- passer par le menu
Tools-> Install Packages ...et sélectionner un site miroir du CRAN - Aller dans l’onglet Packages dans la fenêtre en bas à droite et cliquer sur Install.
- utiliser la fonction
install.packages()directement dans la console - télécharger l’archive .zip correspondant au package et utiliser
ensuite
Tools-> Install Packages ..., choisirPackage Archive File (.tar,.zip)dans Install form.
- installer directement dans un terminal (sans utiliser RStudio) avec
la commande
R CMD INSTALL package-name.tar.gz.
Attention, vous devez posséder une version de R qui permette l’installation du package désiré et certains packages nécessitent l’installation préalable d’autres packages.
Pour l’installation d’un package de Bioconductor, il faut d’abord
installer BiocManager
install.packages("BiocManager") puis on utilise la commande
BiocManager::install().
Pour installer une librairie hébergée sur un dépot github, il faut
d’abord installer le package devtools
install.packages("devtools"), charger cette librairie
library(devtools) puis utiliser la commande
install_github().
Toutes les librairies ne sont pas chargées automatiquement au
lancement de R. Pour charger une librairie, on utilise la commande
library() et les commandes search() ou
searchpaths() pour connaître la liste des librairies
chargées. Vous pouvez utiliser la commande .libPaths() pour
connaitre les chemins vers lesquels pointe R pour trouver des librairies
installées. Il faut aussi parfois mettre à jour les librairies. Pour
cela, vous pouvez aller dans l’onglet Packages dans la
fenêtre en bas à droite et cliquer sur l’icône Update,
ou en utilisant la commande update.packages(...).
Exercice
Enoncé
- Installer la dernière version de R et RStudio
- Installer les packages suivants qui nous serviront par la suite : tidyverse, FactoMineR, mixOmics, corrplot, gridExtra, plotly, questionr
- Charger la librairie ggplot2, la librairie
tidyverse. Y-a-t-il un lien entre les deux ?
- Accéder à la documentation de tidyverse, de la fonction rnorm.
Correction
Question 1 : Voici pour les trois premiers, faire de même pour les autres.
install.packages("FactoMineR")
install.packages("tidyverse")
install.packages("BiocManager")
BiocManager::install("mixOmics")Question 2 :
library(ggplot2)
library(tidyverse)Le package tidyverse englobe plusieurs librairies dont ggplot2. On reviendra sur ce package tidyverse dans le tutoriel 4 de R avancé.
Question 3 :
help(tidyverse)
`?`(tidyverse)
help(rnorm)
`?`(rnorm)